Texto feito por Maurílio Bonora Junior e Ana Arnt
A reabertura em muitos estados do Brasil já é uma realidade. Mesmo com muitos especialistas aconselhando contra isso. Assim, independente de ainda ser prudente que parte da população ainda permaneça em casa, as pessoas têm aderido cada vez menos às medidas de isolamento social. Bem como, infelizmente, cada vez menos se vê o uso de máscaras em espaços públicos. Apesar disso, também vemos vários indivíduos temerosos com a “volta à normalidade”. Em nosso cotidiano, temos recebido muitos questionamentos acerca de qual tipo de máscaras usar, e quais testes fazer em casos de sintomas de gripe e suspeita de Covid-19. Pois bem, estamos aqui para te ajudar!
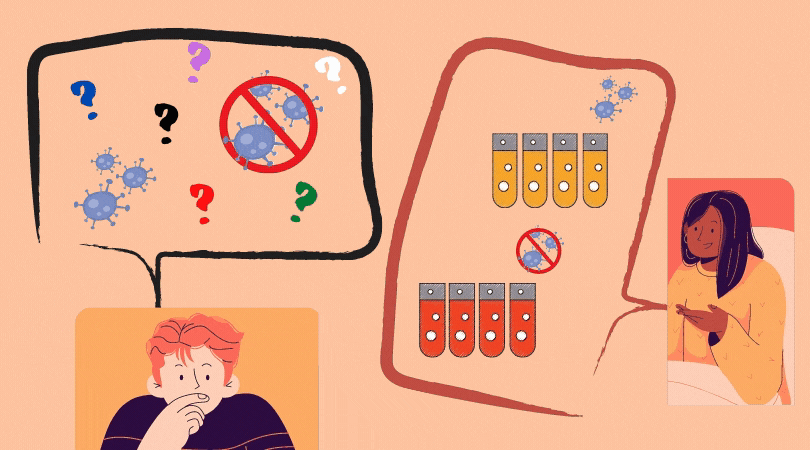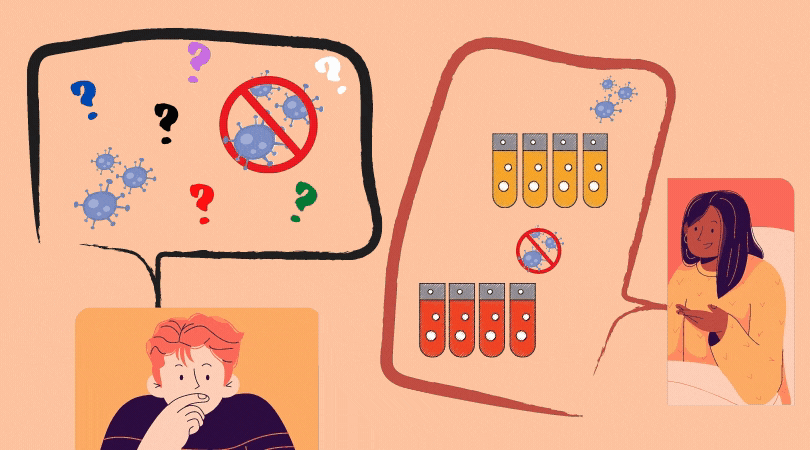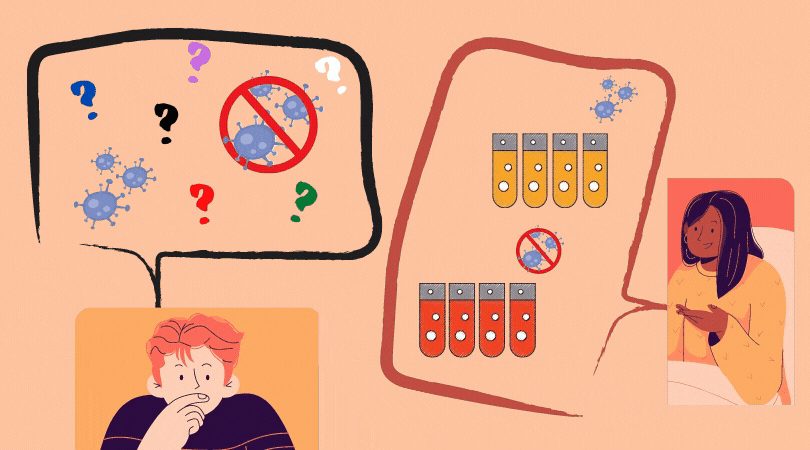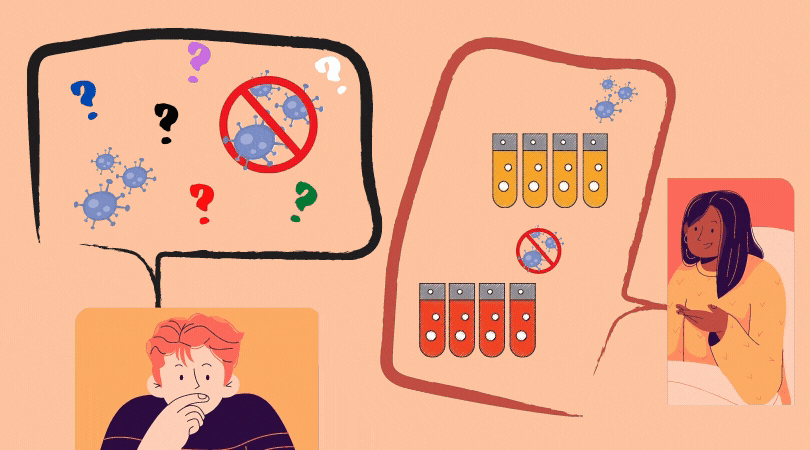
Qual a diferença entre todos esses testes?
Testes RT-qPCR:
Como já falamos dele em um post inteirinho há um tempo atrás, aqui no Especial, vamos só relembrar algumas coisas agora. Esse teste se baseia em detectar o material genético do SARS-CoV-2, o vírus causador da Covid-19, na nossa garganta ou nariz. Para isso, os profissionais de saúde passam um cotonete (chamado de swab) dentro do nosso nariz e garganta. Depois disso, encaminham esse material para um laboratório, onde esse material irá ser utilizado para a detecção do vírus.
O importante aqui é saber que tal teste é muito sensível, isso é, ele consegue detectar baixas quantidades de vírus nas nossas mucosas. Por causa disso, ele é mais indicado para o diagnóstico da Covid-19. Isso é importante pois, utilizando esse teste, é possível saber se uma pessoa assintomática está com o vírus ou não. Sendo, portanto, uma possível fonte de transmissão mesmo sem ter tal conhecimento.
Testes sorológicos:
Este é outro teste de laboratório. Ele utiliza produtos do nosso próprio corpo para saber se estamos com uma infecção de Covid-19 ou se já tivemos a doença: os anticorpos. Nesse tipo de teste, profissionais da saúde colhem uma amostra de sangue da pessoa. Após a coleta testam com amostras do patógeno, buscando identificar se o sangue possui anticorpos contra este, no caso, o vírus da Covid-19. Lembrando que também fizemos um texto falando sobre anticorpos e como são produzidos. Nesse teste, busca-se identificar dois tipos específicos de anticorpos, os IgM e IgG. É a partir da combinação de presença e ausência desses anticorpos que se tem o resultado:
- Presença somente de IgM: a pessoa está no momento ou esteve com uma infecção de Covid-19 recentemente;
- Quando há presença somente de IgG: a pessoa entrou em contato com o vírus da Covid-19 no passado;
- Presença de IgM e IgG: a pessoa está no momento com uma infecção prolongada (crônica) de Covid-19;
- Ausência de ambos: a pessoa nunca teve Covid-19 OU apesar de já ter tido Covid-19 têm poucos anticorpos no sangue OU está em um período inicial da infecção de Covid-19 e não teve tempo de produzir anticorpos, ainda.
Um ponto importante desse tipo de teste é que ele detecta de forma indireta a presença do vírus no organismo. Ou seja, os anticorpos que o corpo produziu devido a infecção. Além disso, é importante lembrar que os anticorpos que produzimos durante uma infecção (especialmente o IgG, mas também o IgM) podem durar por semanas e até meses. Dessa forma, não se indica tais testes para se diagnosticar se uma pessoa está com Covid-19.
Testes Rápidos:
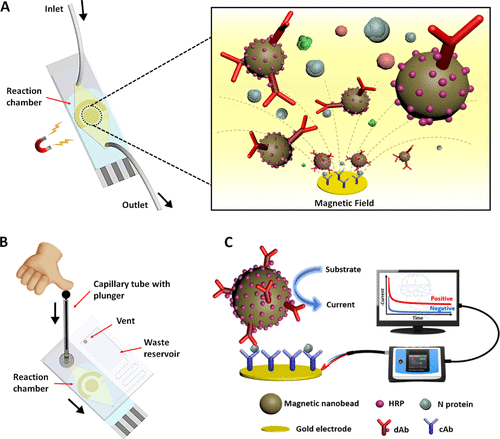
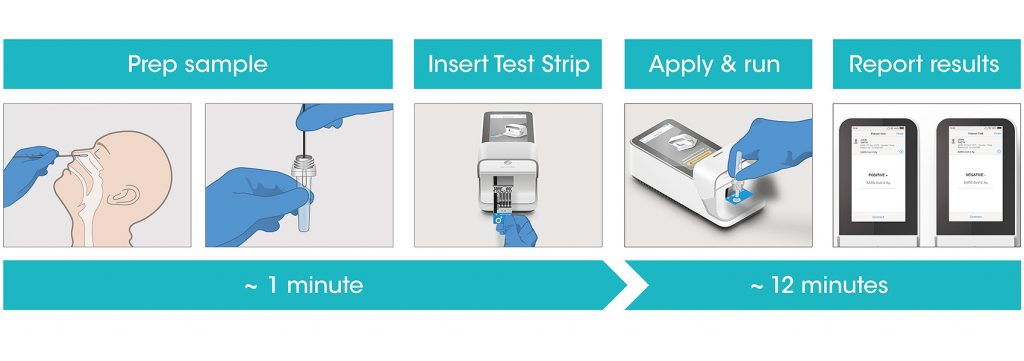
São os famosos “testes de farmácia”. Fisicamente parecidos com os testes de gravidez, foram bastante comentados na mídia durante os primeiros meses de pandemia. Todavia, agora, com a reabertura prematura dos comércios, escolas, e sociedade como um todo ressurgiram como pauta. Esse tipo de teste se baseia em uma técnica chamada cromatografia. Em termos mais simples, eles possuem uma substância chamada de reagente ou sensor, que vai reconhecer os anticorpos do nosso sangue (os Testes Rápidos de Anticorpo) ou uma parte específica do vírus, a partir da nossa saliva ou secreção do nariz (nos Testes Rápidos de Antígeno). Quando essa substância se liga aos anticorpos ou ao vírus, acontece uma série de reações químicas que fazem com que ela mude de cor. Essa mudança de cor aparece como pequenas faixas coloridas no teste, apontando a presença ou ausência dos anticorpos contra o SARS-CoV-2, ou do próprio vírus.
Testes Rápidos de Anticorpo e de Antígeno: algumas diferenças
A leitura do resultado do teste rápido de anticorpo parece com o do teste sorológico. Uma vez que aponta a presença ou ausência de IgM e IgG. Já a leitura do teste rápido de antígeno é mais simples. Pois este aponta somente a presença ou ausência da proteína Spike do SARS-CoV-2 na nossa mucosa. O fato interessante é que ambos os testes também tem uma marcação chamada de “C”, que quer dizer o controle. Caso não apareça um risco nessa marcação, é um sinal de que o teste não funcionou corretamente. Dessa maneira, aquele resultado não é confiável, sendo necessário refazê-lo ou realizar outro teste (sorológico ou RT-qPCR).
Assim como os testes sorológicos, os testes rápidos de anticorpos detectam a presença do vírus da Covid-19 de forma indireta, via os anticorpos que produzimos. Enquanto isso, os testes rápidos de antígeno detectam o vírus diretamente. Os pontos positivos aqui são que o resultado sai muito mais rápido do que nos outros dois tipos de teste. Uma vez que saibamos a resposta em pouco minutos (entre 15 e 30 minutos). Além disso, esse teste custa mais barato comparado aos outros, podendo-se realizar na própria farmácia. Contudo, temos um grande ponto negativo: a sua sensibilidade.
A sensibilidade de um teste se refere a capacidade deste de detectar quantas pessoas realmente com anticorpos ou com o vírus tiveram um teste positivo. Exemplo:
“Em um teste rápido de anticorpos com sensibilidade de 75%, a cada 100 pessoas que realmente tenham o anticorpo contra o vírus da Covid-19, 75 dessas pessoas terão um resultado positivo (chamado de verdadeiro positivo), e as outras 25, mesmo que tenham o anticorpo no sangue, terão um resultado negativo (chamado de falso-negativo).”
Em outras palavras, mesmo se eu estivesse com o vírus e tivesse anticorpos contra ele no meu sangue, nesse exemplo, eu teria 25% de chance de ter um resultado errado. Ou seja, apontando que não estava contaminado.
Um outro complicador desse teste é a janela de tempo em que ele pode ser realizado. No caso dos testes rápidos de anticorpos, é aconselhado fazer este somente após o 8º dia de sintomas. Uma vez que seria somente a partir desse período de tempo que teríamos anticorpos suficientes no sangue para serem detectados pelo exame. Já no caso do teste rápido de antígeno, eles são indicados para serem feitos entre o 2º e 7º dia de sintomas. Isto é, quando há uma quantidade grande de vírus na mucosa.
Entretanto, aqui é necessário fazer algumas ressalvas:
- Mesmo fazendo esse tipo de teste na janela de tempo correta, o resultado ainda pode ter margem de erro por causa da sensibilidade dele (explicada acima).
- Esses testes precisam ser feitos, analisados e interpretados por profissionais de saúde treinados, e não por qualquer pessoa
- De forma alguma, para diagnóstico da COVID-19, indica-se os testes rápidos
Mas enfim, devo ou não fazer o teste rápido?
Muitos planos de retomada, como o do estado de São Paulo, indicam (vagamente) esses tipos de testes (sorológicos e rápidos) como forma de diagnóstico em casos suspeitos. Entretanto, nossa indicação resposta curta ainda é:
Não, eles não podem ser utilizados para o diagnóstico.
Como forma de diagnosticar a Covid-19, não se indica os testes rápidos de antígeno ou anticorpo, tanto quanto os sorológicos. Assim, a testagem por RT-qPCR, como indicado pela OMS, ainda é a recomendada. Uma vez que este teste tem uma sensibilidade muito maior que os outros testes, quando feito no período certo (entre o 4º e 12º dia). Isto é, o teste de RT-qPCR tem uma taxa de sucesso muito maior do que os outros. Pois, pode identificar inclusive casos assintomáticos da doença com muito mais facilidade.
Entretanto, não é como se os testes rápidos não servissem para nada. Conforme indicado pelo FDA, como forma de monitorar pessoas os testes rápidos são bastante indicados por serem uma solução prática, eficiente e barata, em situações específicas. Quais? Pessoas em constante contato com pacientes de Covid-19, de grupo de risco, como médicos, enfermeiros, trabalhadores em lares de idosos, etc. Além disso, mesmo para rastreio de contatos ou testes por amostras em grupos, dentro de grandes empresas, estes testes podem ser ferramentas eficientes.
Eu fiz o teste rápido de antígeno e deu negativo, mas estou com sintomas de COVID-19, como proceder?
Quando houver suspeita de estarmos com COVID-19 a recomendação sempre é realizar o teste de PCR. Caso isso não seja uma possibilidade e a saída for um teste rápido de antígeno, independente do resultado, reforça-se a necessidade de afastamento por 14 dias após os sintomas desaparecerem (em especial tosse e febre).
Em ambientes de trabalho em que as pessoas permaneçam um longo período de tempo juntas, quando acontece o contato com alguém positivado, a indicação é de teste de todas as pessoas que estiveram contato e de afastamento por 10 dias na ausência de sintomas. Caso apareçam sintomas, a indicação é refazer o teste e manter-se afastado por 14 dias após os sintomas de febre e tosse desaparecerem.
Sobre o tempo de incubação da COVID-19, escrevemos um texto em 2020, cujos tempos seguem válidos. Ainda ressaltamos que mesmo vacinados as pessoas devem seguir os protocolos de uso de máscara (preferencialmente PFF2), distanciamento físico / social, baixa permanência em ambientes fechados e mal ventilados.
Mas quais sintomas eu devo levar em consideração?
Sintomas gripais em geral, tosse seca, febre e cansaço estão entre os sintomas mais comuns, seguidos por dor de cabeça, diarréia, perda de olfato e/ou paladar e dor de garganta. Os sintomas graves que devem ter procura de unidades de saúde são falta de ar, dificuldade de respirar e dor no peito.
Finalizando
Sintomas gripais, em tempos de COVID-19 não só podem mas devem ser entendidos como suspeito de COVID-19 e ter afastamento imediato, sem ônus (salarial, de faltas no trabalho e/ou em aulas) para as pessoas. Mesmo com a vacinação aumentando em quantidade diariamente, ainda temos uma alta transmissão e não devemos negligenciar as medidas sanitárias mais simples, além da indicação de teste e rastreio para todas as idades.
REFERÊNCIAS:
- Flores, MG, Diniz, LP, Shiomatsu, GY, Ninomiya, VY, Carvalho, RT AFINAL, COMO FUNCIONA O TESTE RÁPIDO PARA COVID-19? Blog Coronavírus Secretaria de Estado de Saúde de Minas Gerais
- Centerlab, TESTE RÁPIDO PARA COVID-19 AG ANTÍGENO ORAL EM AMOSTRA DE SALIVA.
- FDA, EUA Authorized Serology Test Performance
- Harmon, A, Chang, C, Salcedo, N, Sena, B, Herrera, BB, Bosch, I, & Holberger, LE (2021) Validation of an At-Home Direct Antigen Rapid Test for COVID-19 JAMA Network Open, 4(8), e2126931-e2126931
- CDC, Interim Guidance for Antigen Testing for SARS-CoV-2
- CDC Overview of Testing for SARS-CoV-2 (COVID-19)
- COVID-19: Diagnosis
Os autores
Maurílio Bonora Junior é biólogo, mestre e doutorando em Genética e Biologia Molecular pela Unicamp, membro da equipe científica do Especial COVID-19.
Ana Arnt é bióloga, doutora em Educação, Professora do Instituto de Biologia da Unicamp e coordenadora do Blogs de Ciência da Unicamp e do Especial COVID-19.
Este texto é original e escrito com exclusividade para o Especial Covid-19