Por: Rafael Sanchez Luperini e Renato Augusto Corrêa dos Santos
PCR, qPCR, RT-qPCR: o que significam essas siglas e o que elas têm a ver com os fungos? As discussões sobre técnicas e metodologias científicas utilizadas para diagnóstico da COVID-19 se popularizaram nos mais diversos meios de comunicação.
O RT-qPCR (Reverse Transcriptase Quantitative Polymerase Chain Reaction, em inglês), por exemplo, é o método mais eficaz para diagnosticar um paciente e ficou conhecido como “o teste do cotonete no nariz”. E nós queremos saber:
- Você já se perguntou como ele funciona?
- Como e quando surgiu essa metodologia científica tão avançada?
- Você sabia que existem ainda outras variantes desta técnica, chamadas de PCR, qPCR e RT-PCR?
- Além das letras em cada sigla, quais as verdadeiras diferenças por trás de cada uma dessas técnicas?
Esse texto busca trazer as respostas para quem está cheio de dúvidas a respeito dessas interessantes, e extremamente versáteis, ferramentas das ciências biológicas.
A história do PCR
Essas metodologias são geralmente aplicadas na identificação de seres vivos a níveis bastante específicos, e para esclarecer melhor todas essas perguntas, vamos explicar a técnica, juntamente com a sua história.
Em 1983 aconteceu uma das mais significantes descobertas do século XX. O cientista Dr. Kary Mullis desenvolveu a reação em cadeia da polimerase (Polymerase Chain Reaction ou PCR). A partir dessa técnica se tornou possível obter muitas cópias de um mesmo fragmento de material genético, possibilitando a obtenção de grandes quantidades de DNA de uma amostra genética de um organismo.
A técnica possibilita a produção de fragmentos de DNA de interesse partindo de pequenas quantidades de amostras de DNA usando a enzima DNA polimerase, a mesma que participa da multiplicação do material genético nas células. Esta enzima se liga a um pequeno fragmento (o iniciador, ou primer, em inglês), desenhado especialmente para se ligar ao DNA alvo, produzindo uma sequência complementar ao fragmento de DNA de interesse, escolhido antes do início da análise.
O primeiro estudo detalhando a metodologia da técnica foi publicado no periódico científico Science, no ano de 1985, revolucionando a ciência e as possibilidades de descobertas ao se trabalhar com DNA. Porém essa metodologia ainda apresentava uma série de desafios, visto que é composta de 3 etapas demonstradas na imagem abaixo:
Reação em cadeia da polimerase explicada passo a passo
A realização de 20 a 40 ciclos promove a amplificação da região que se pretende analisar, seja ela um gene humano específico, ou de microrganismos ou basicamente qualquer material genético que se deseja multiplicar para analisar posteriormente.
Variações da técnica de PCR
Ao longo dos anos, começaram a surgir variações da técnica, e aplicações das mais diversas formas, e é nesse contexto que surge a análise tão utilizada hoje em dia nos diagnósticos de COVID-19, a RT-qPCR.
E o que significam todas essas letras adicionadas antes da PCR?
Elas dizem respeito a uma metodologia com uso de uma enzima chamada Transcriptase Reversa (Reverse Transcriptase, ou RT), que tem a função de produzir uma fita de DNA (chamada de DNA complementar ou cDNA) a partir de uma fita de RNA.
Além disso, a letra “q” indica que esta técnica é quantitativa e pode ser usada em RT-qPCR e qPCR. Esta metodologia se parece muito com a PCR original, porém com a diferença de que são adicionadas sondas fluorescentes de DNA junto das amostras, as quais emitem fluorescência a cada ciclo realizado pelo aparelho.
Portanto, durante a amplificação, a quantificação de DNA é determinada pela quantidade de fluorescência emitida pelo produto amplificado a cada ciclo.
Isso é possível somente com a utilização de um sistema de equipamentos com monitoramento da fluorescência emitida, possibilitando uma quantificação mais exata de quanto material genético existia na amostra inicial, abrindo ainda mais opções e oportunidades de análises a serem feitas, como será explicado a seguir.
Pesquisas que utilizam a técnica PCR
Agora vamos dar alguns exemplos de pesquisas importantes no Brasil e ao redor do mundo, que utilizam a técnica de PCR e suas variantes.
Aplicações de PCR nas pesquisas agrícolas do Brasil.
Além dos interesses das aplicações da técnica na área da saúde, a agricultura e a indústria de alimentos e bebidas também se beneficiam da técnica. Em algumas situações, a qPCR é utilizada em laboratórios de análise de alimentos visto que é uma técnica altamente específica e sensível. Porém, dentre as dificuldades estão seu alto custo devido à necessidade de mão de obra especializada, insumos e metodologia para a detecção e identificação de determinados microrganismos.
Diversas pesquisas desenvolvidas no Brasil visam o melhoramento da detecção de fungos que contaminam alimentos, como é o caso de espécies das espécies Aspergillus niger e Aspergillus welwitschiae, produtoras de micotoxinas, algumas delas nefrotóxicas e potencialmente carcinogênicas.
Pesquisas na Universidade Estadual de Campinas (UNICAMP) em 2018, coordenadas pela Dra. Marta Hiromi Taniwaki e em colaboração com pesquisadores da Universidade Estadual de Londrina (UEL) estudaram o uso da técnica de qPCR na detecção destas espécies citadas acima, obtidas de café. O método desenvolvido possibilita rápida, precisa e sensível detecção das espécies citadas, que são morfologicamente idênticas.
PCR em laboratórios de fitopatologia
No Brasil, há algumas clínicas fitopatológicas que fazem a análise de qPCR para a detecção de doenças importantes em plantas, como é o caso da EMBRAPA, a ESALQ (Universidade de São Paulo, em Piracicaba), o Centro de Cana e o Centro de Citricultura, ambos pertencentes ao Instituto Agronômico de Campinas (IAC).
A pesquisadora Laís Moreira Granato, do Instituto Agronômico de Campinas (IAC), contou um pouco sobre a aplicação de qPCR no cotidiano. Na prática, laboratórios de fitopatologia usam a técnica de RT-qPCR para a detecção de vírus de RNA e atécnica de qPCR para detecção de fungos e bactérias em citros. Geralmente o citricultor leva amostras de frutas ou folhas para a clínica do Centro de Citricultura procurando por essas doenças.
Infelizmente, como ja foi dito, os insumos e equipamentos que envolvem a qPCR são caros! No entanto, os citricultores precisam desse serviço para ter certeza de que não há doenças escondidas em seus pomares.
Uma das motivações para se pagar um pouco mais por esse serviço envolve a legislação que regula a exportação dos vegetais. Frutas de mesa, quando exportadas para a Europa, precisam obrigatoriamente passar por uma comprovação de que não há presença de alguns fungos. Um exemplo é Phyllosticta citricarpa nas cascas das laranjas. Este fungo não existe na Europa e a legislação não permite que nada entre sem uma comprovação de que está “limpo”.
Na prática, em algumas situações, mesmo que a detecção pudesse ser feita, questões ligadas ao sistema produtivo podem ser um problema, como a limitação de equipamentos, disponibilidade de equipes de inspeção e de corpo técnico. Mas, além disso, descobrimos algo curioso quando o assunto é priorizar um problema ou outro na agricultura, e que podem deixar os fungos “de lado”, como nos contou a pesquisadora Dra. Andressa Bini, do Centro de Cana do IAC.
O exemplo é o fungo Colletotrichum falcatum, causador da podridão vermelha em cana-de-açúcar. Acreditava-se que o fungo infectaria apenas plantas a partir de ferimentos causados por uma praga, a lagarta de Diatraea saccharalis. Seguindo este raciocínio, a prioridade no passado era controlar apenas a praga, mas não o fungo em si, que seria uma consequência oportunista.
No entanto, a realidade é que os fungos conseguem infectar as plantas mesmo na ausência da praga chamada de “broca”, tornando a detecção do fungo uma prioridade, já que sem um controle efetivo da doença, podem ocorrer perdas de até 35% da produção e hoje o patógeno já ocorre pelo menos no Triângulo Mineiro, no Mato Grosso do Sul e em algumas regiões de São Paulo.
Os eucaliptos e o fungo Austropuccinia psidii
Outro exemplo de pesquisa aplicada e com uso de qPCR também vem da ESALQ! O eucalipto é uma planta muito importante para a produção de madeira e papel em nosso país. Uma doença fúngica causada por Austropuccinia psidii, a ferrugem, é conhecida como problemática para esta cultura.
Um grande problema da detecção desta doença é que o fungo é normalmente percebido apenas após o aparecimento de sintomas nas plântulas, quando o problema já é muito grande. Os métodos usados geralmente são pouco eficientes ou pouco sensíveis. Apostando na qPCR, mais sensível, mais rápida e menos laboriosa, pesquisadores da Universidade de São Paulo (USP), em 2018, propuseram o uso de qPCR para a detecção prematura da doença em eucalipto. Outra aplicação interessante desta análise, sugerida pelos pesquisadores, é a identificação rápida de plântulas suscetíveis ou resistentes à doença em programas de melhoramento.
Como pudemos ver, geralmente as limitações ainda estão no alto custo dessa tecnologia recente, porém as aplicações são as mais diversas, e ainda há muito para se desenvolver na área. Ainda estamos no começo de uma nova era, e a tendência é que a técnica seja aprimorada e torne-se mais barata e aplicável com o passar dos anos.
Fontes consultadas
- Dra. Laís Moreira Granato (Instituto Agronômico de Campinas – IAC)
- Dra. Andressa Peres Bini (Centro de Cana – IAC)
- Dra. Daniele Sartori (Universidade Estadual de Londrina, UEL)
- Artigo científico intitulado “A Real Time PCR strategy for the detection and quantification of Candida albicans in human blood.”, publicado na revista do Instituto de Medicina Tropical de São Paulo 62 em 2020, de autoria de Busser, F. e colaboradores.
- Artigo científico intitulado “A New Age in Molecular Diagnostics for Invasive Fungal Disease: Are We Ready?”, publicado na revista Frontiers in Microbiology em 2020, de autoria de Kidd, S. e colaboradores.
- Artigo científico intitulado “Development of a quantitative real-time PCR assay using SYBR Green for early detection and quantification of Austropuccinia psidii in Eucalyptus grandis.” publicado na revista European Journal of Plant Pathology 150.3 em 2018, de autoria de Bini, A. e colaboradores.
- Artigo científico intitulado “Real-time PCR-based method for rapid detection of Aspergillus niger and Aspergillus welwitschiae isolated from coffee.” publicado na revista Journal of microbiological methods 148 em 2018, de autoria de Von Hertwig, A. e colaboradores.
- Matéria no site da empresa Kasvi intitulada “História e evolução da técnica de PCR (Polymerase Chain Reaction ou Reação em Cadeia da Polimerase)” publicada em 18/06/2015. (Website).
- Matéria no site da empresa Kasvi intitulada “Qual a diferença entre PCR e qPCR?” publicada em 30/04/2015. (Website).
- Matéria no Blog Biomedicina Padrão intitulada “A evolução da PCR” publicada em 05/12/2013. (Website).
- Matéria no Blog Biomedicina Padrão intitulada “Reação em Cadeia da Polimerase – PCR” publicada em 14/06/2020. (Website)
- Site da Embrapa (Website)
Sobre os autores
Rafael Sanchez Luperini é aluno de pós-graduação (mestrado) pelo programa de Bioquímica da Faculdade de Medicina de Ribeirão Preto (FMRP) na Universidade de São Paulo (USP), atualmente orientado pelo Prof. Dr. Gustavo H. Goldman (FCFRP, USP Ribeirão Preto). Trabalha com espécies do gênero Aspergillus, buscando desvendar as diferenças entre espécies de fungos.
CV Lattes: http://lattes.cnpq.br/7815439327487936
E-mail: rafaluperini@gmail.com
Instagram: @rafasluperini
Facebook: https://www.facebook.com/rafaluperini/
Renato Augusto Corrêa dos Santos é doutorando pelo programa de Genética e Biologia Molecular da Universidade Estadual de Campinas (UNICAMP), fazendo análises genômicas de fungos patogênicos do gênero Aspergillus, sob orientação do Prof. Dr. Gustavo H. Goldman (FCFRP, USP Ribeirão Preto) e com financiado da FAPESP. Seu projeto envolve uma colaboração do com o LGE (UNICAMP) e o Rokas Lab (Vanderbilt University, EUA).
CV Lattes: http://lattes.cnpq.br/3339727232509001
E-mail: renatoacsantos@gmail.com
Instagram: @renato.correa.182
Facebook: https://www.facebook.com/renato.correa.182
Este texto foi escrito com originalmente no Blog Descascando a Ciência

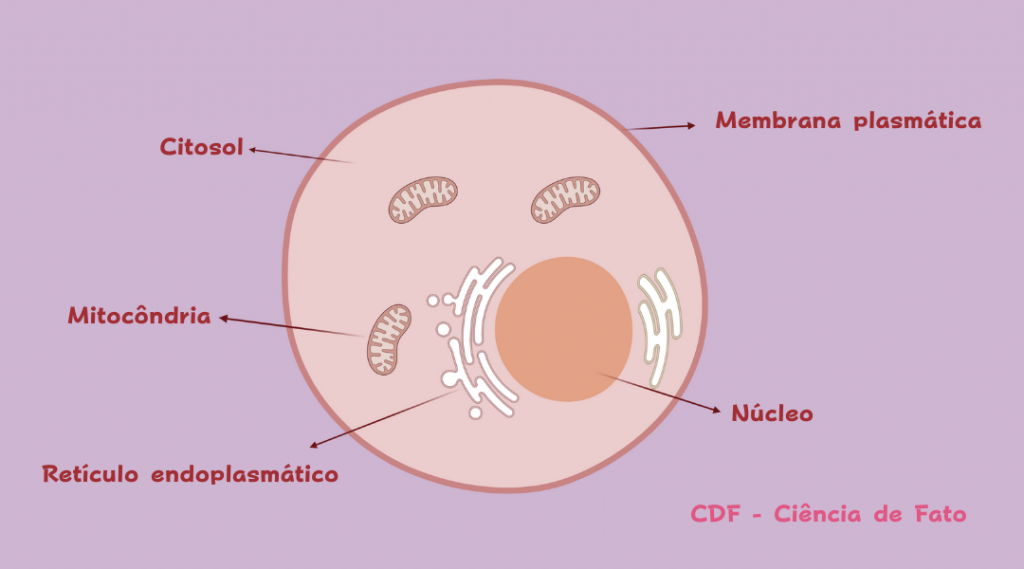
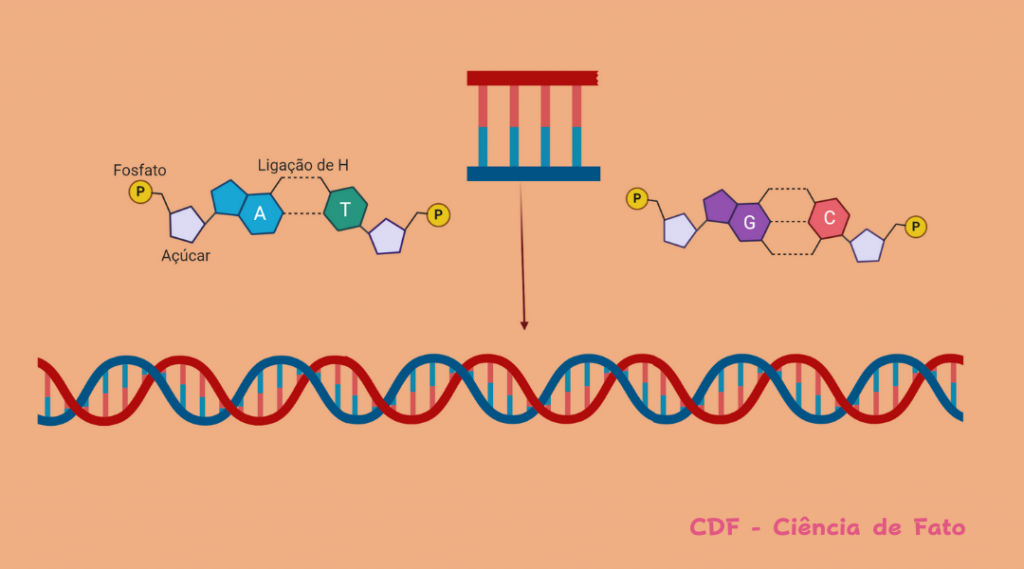
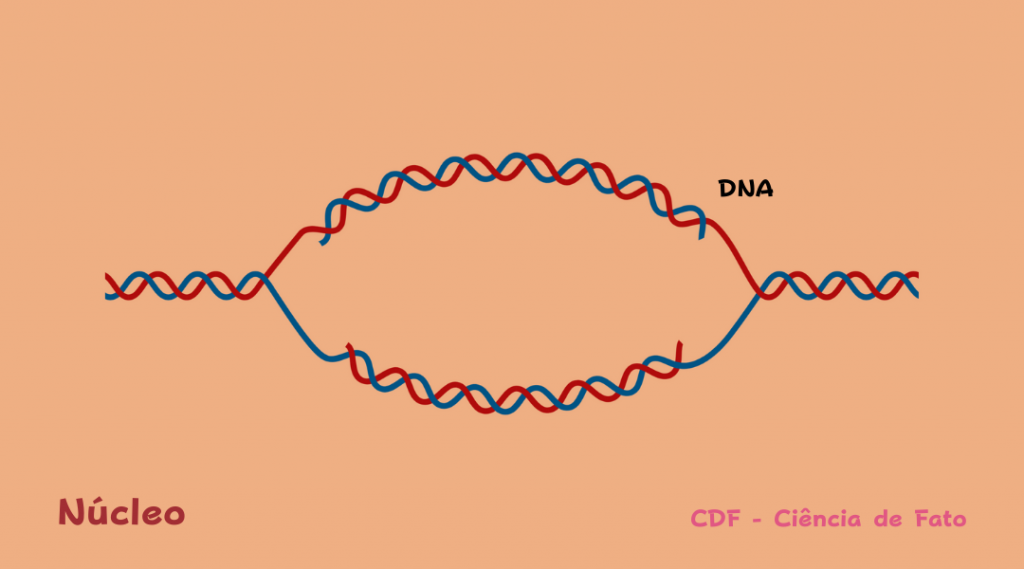
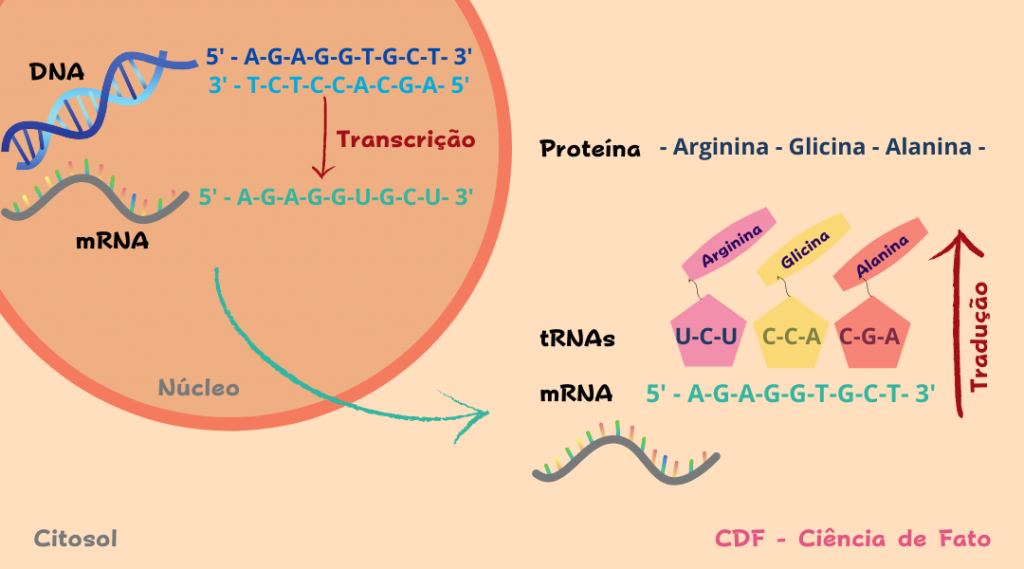


 Como nosso genoma integra sinais intrínsecos e ambientais sem que haja alteração da sequência de DNA? Você já parou para pensar qual o impacto de seus hábitos sobre sua vida? E se as experiências vividas pudessem ser transmitir aos filhos? Qual seria o impacto de tudo isso? Hoje se sabe que as informações contidas em nosso genótipo não possuem controle exclusivo sobre nossa identidade. Sendo assim, a expressão dos genes contidos em nossas células é mediada por diversos fatores em que a informação não está contida apenas no alfabeto do DNA (ATGC).
Como nosso genoma integra sinais intrínsecos e ambientais sem que haja alteração da sequência de DNA? Você já parou para pensar qual o impacto de seus hábitos sobre sua vida? E se as experiências vividas pudessem ser transmitir aos filhos? Qual seria o impacto de tudo isso? Hoje se sabe que as informações contidas em nosso genótipo não possuem controle exclusivo sobre nossa identidade. Sendo assim, a expressão dos genes contidos em nossas células é mediada por diversos fatores em que a informação não está contida apenas no alfabeto do DNA (ATGC).